
今天推送的文章来自于2021年11月发表在ACS Catalysis上的“Binding Affinity Determines Substrate Specificity and Enables Discovery of Substrates for N-Myristoyltransferases”。通讯作者为美国康奈尔大学的 Hening Lin 教授。
kcat/Km被认为是酶对其底物催化效率或底物特异性的量度。具有较高kcat/Km值的底物被认为是更好或优选的底物。N-肉豆蔻酰转移酶 (NMT) 催化许多真核蛋白质的 N 端甘氨酸肉豆蔻酰化。在研究人 N-肉豆蔻酰基转移酶 1 (NMT1) 时,本文作者意外地发现 NMT1 对具有相似kcat/Km值的两种底物表现出截然不同的偏好,导致当两种底物都存在于反应中时,只有一种底物发生周转。这一观察结果表明结合亲和力是比kcat/Km更有效的底物特异性预测因子。此外,作者证明了基于结合亲和力的方法可以轻松识别控制蛋白质翻译后修饰 (PTM) 的酶的底物蛋白质。
N-末端甘氨酸肉豆蔻酰化是真核细胞中由 N-肉豆蔻酰基转移酶 (NMT) 催化的重要且进化上保守的 PTM。它调节对广泛生物过程至关重要的许多人类蛋白质的膜靶向、蛋白质稳定性和蛋白质-蛋白质相互作用。人的两种NMT,NMT1和NMT2,对肉豆蔻酰辅酶A和蛋白质N-端甘氨酸具有很高的选择性。但是,当作者使用 ARF6 N-端肽 (GKVLSKIFWW) 测试重组人 NMT1 时,乙酰辅酶 A 也可以用作底物,导致乙酰肽产物的形成。与不含 NMT1 的相应对照反应相比,在含有 ARF6 肽、肉豆蔻酰辅酶 A 和 NMT1 的反应中检测到肉豆蔻酰化ARF6肽;而在含有 ARF6 肽、乙酰辅酶 A 和 NMT1 的反应中,也观察到乙酰化 ARF6 肽的产生。因为众所周知 NMT1 选择性地使用肉豆蔻酰辅酶 A 作为底物,所以观察到它可以在体外催化乙酰化是令人惊讶的。因此,作者假设在体内,只要肉豆蔻酰辅酶 A的kcat/Km值远高于乙酰辅酶 A 的,NMT1 仍然可以主要催化肉豆蔻酰化。因此,测量了 NMT1 催化 ARF6 肽乙酰化和肉豆蔻酰化的稳态动力学参数。出乎意料的是,kca和Km值非常相似。


如果kcat/Km值决定了底物特异性,那么 NMT1 应该能够催化细胞中 ARF6 的乙酰化和肉豆蔻酰化。然而,对从细胞中分离的 ARF6 进行分离表征后并没发现任何乙酰化的 ARF6。因此,在这种情况下,kcat/Km值具有误导性。为了进一步研究这一点,作者使用 200 μM ARF6 肽建立了 NMT1 反应,并添加了 50 μM 的肉豆蔻酰辅酶 A 和乙酰辅酶 A。在该反应中,检测到约 12 μM 肉豆蔻酰化的 ARF6 肽,没有乙酰辅酶 A 的反应。因此,尽管kcat/Km值相似,但 NMT1 强烈偏好肉豆蔻酰辅酶 A 而非乙酰辅酶 A。

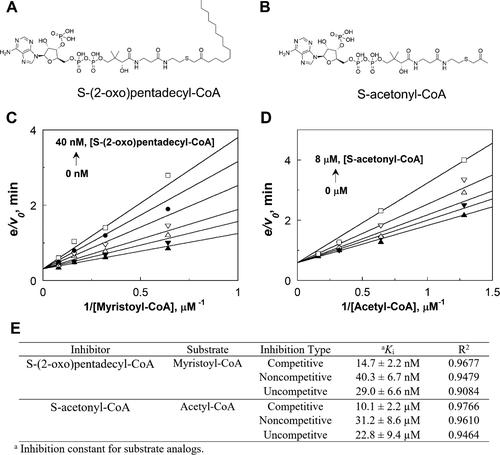
为了解释为什么当肉豆蔻酰辅酶 A 和乙酰辅酶 A 都存在时 NMT1 几乎只使用肉豆蔻酰辅酶 A 作为底物,作者推测 NMT1 与肉豆蔻酰辅酶 A 的结合比它与乙酰辅酶 A 结合强,因此,当两者都存在于反应中时,NMT1 将几乎完全被肉豆蔻酰辅酶 A 占据并仅催化肉豆蔻酰化。为了验证这一假设,作者开始测量 NMT1 对肉豆蔻酰辅酶 A 和乙酰辅酶 A 的结合亲和力。因为从大肠杆菌中表达和纯化的人 NMT1已经与肉豆蔻酰辅酶 A 紧密结合,所以直接测量K d不可行。在作者以前发表的 NMT1 与肉豆蔻酰化 ARF6 肽结合的结构中,作者发现即使没有添加肉豆蔻酰辅酶 A,在晶体中也观察到辅酶 A 的电子密度,这再次表明 NMT1 与肉豆蔻酰辅酶 A 非常紧密地结合,而纯化的 NMT1 已经与肉豆蔻酰辅酶 A 结合. 透析去除结合的肉豆蔻酰辅酶 A 也没有成功。于是作者合成了肉豆蔻酰辅酶 A 类似物 S-(2-氧代)十五烷基辅酶 A 和乙酰辅酶 A 类似物 S-丙酮酰辅酶 A,在 CoA 硫和酰基羰基之间引入亚甲基桥使这两种类似物不可被水解。然后研究了 S-(2-氧代)十五烷基辅酶 A 抑制 NMT1 肉豆蔻酰基转移酶活性的能力和 S-丙酮酰辅酶 A 抑制 NMT1 乙酰转移酶活性的能力。考虑到底物类似物和底物之间的高度结构相似性,S-(2-氧代)十五烷基辅酶A 和 S-丙酮酰辅酶A的K i值分别反映了 NMT1 对肉豆蔻酰基辅酶A和乙酰辅酶A的结合亲和力。结果表明S-(2-氧代)十五烷基辅酶A 的K i值约为15 nM,S-丙酮酰辅酶A的K i值为10.1 ± 2.2 μM,比S-(2-氧代)十五烷基辅酶A 大近3个数量级,转换成对应的结合亲和力后,这种巨大差异就解释了 NMT1 对肉豆蔻酰辅酶 A 的特殊偏好。
根据kcat/Km决定底物特异性的传统观点,一种酶与其优选的底物不一定具有很强的结合亲和力。相反,他们只需要有更高的kcat/Km值。然而,在Km=Kd的情况下,kcat/Km仍然是一个有效的特异性常数,作者想强调的是,在更复杂的生理条件下,存在具有相似化学和结构特性的多种底物相互竞争酶活性位点时,结合亲和力值应该比kcat/Km更具参考价值。认识到结合亲和力对于确定酶的底物特异性至关重要,这对于旨在了解 PTM 酶生理功能的研究具有相当重要的意义。要了解 PTM 酶的功能,一项重要的任务是确定它们修饰的底物蛋白质。然而,在许多情况下,由于各种原因,例如缺乏适当的亲和富集试剂或底物丰度低,鉴定 PTM 酶的底物蛋白仍然是一个挑战。结合亲和力可能会提供一种简便的通用或补充方法来识别 PTM 酶的底物蛋白——可以寻找与酶相互作用的蛋白质,而不是寻找具有 PTM 的蛋白质。由于当前的蛋白质组学技术在识别相互作用的蛋白质方面非常出色,这将为 PTM 酶底物识别的挑战提供一个简便的解决方案。
为了证明该方法的有效性,作者开始使用 NMT1/2 的现有交互组数据来识别以前未知的 NMT1/2 底物。在与 NMT1 或 NMT2 相互作用的 52 种蛋白质中,有 19 种是 NMT1/2 的已知底物蛋白或已被鉴定为蛋白质组学研究中的潜在底物。这是一个很好的迹象,表明 NMT 相互作用组研究可以导致 NMT 底物蛋白的鉴定。为了从相互作用的蛋白质中识别以前未知的底物蛋白质,作者将注意力集中在未知的 NMT 底物蛋白质上。作者挑选了七个蛋白进行表达,用肉豆蔻酸炔烃 (Alk12) 标记,并在 TAMRA-叠氮化物缀合后通过凝胶内荧光分析它们的脂肪酰化水平。ARMC3和 ODF3L2未显示 Alk12 标记。另外两种蛋白质,PHEAT2和 CADM4 显示出 Alk12 标记信号,但该信号被羟胺处理去除,表明脂肪酰化可能发生在半胱氨酸而不是 N -末端甘氨酸。因此,PHEAT2 和 CADM4 可能不是 NMT1/2 的底物。然而,有三种蛋白质 LRATD1、LRATD2和 ERICH5在 Alk12 处理的样品中显示出清晰的荧光信号,表明它们可能是 N-肉豆蔻酰化的。使用 NMT 抑制剂和 失活突变体G2A 进一步鉴定 LRATD1、LRATD2 和 ERICH5 作为 NMT1/2 底物。Alk12 标记信号随着 LRATD1、LRATD2 和 ERICH5 中 NMT 抑制剂的处理而降低,并在 G2A 突变体中被完全去除,从而确定 LRATD1、LRATD2 和 ERICH5 是 NMT1/2 的底物。因此,通过挖掘可用的 NMT1/2 相互作用组,确定了 NMT1/2 的三种以前未知的底物蛋白。NMT1/2 的三种以前未知的底物的鉴定表明,相互作用组分析可以成为鉴定 PTM 酶底物蛋白的有力方法,特别是考虑到现成的相互作用组数据。

总之,尽管 NMT1 的kcat/Km值相似,但作者对 NMT1 对肉豆蔻酰辅酶 A 偏好优于乙酰辅酶 A 的研究导致人们认识到Kd值是酶底物特异性的关键决定因素。虽然 NMT 的例子可能是Km不等于Kd引起的特殊情况,但作者建议应该更加强调Kd来确定酶的底物特异性,特别是在多个底物蛋白竞争同一个 PTM 的生理环境中酶。该研究表明,对于细胞和体内研究,其中许多底物蛋白竞争相同的 PTM 酶,结合亲和力非常重要,应该更加重视。该思路将有许多实际应用,因为它可以轻松识别 PTM 酶的底物蛋白,这将显著加快对这些酶的生物学功能的理解。


整理:丁孙家
文章信息:
DOI: 10.1021/acscatal.1c03330
,




