SHZ 生信人
哈喽,大家好哇~马上要过年了,小编在这里提前祝您新年快乐,阖家幸福~过年的氛围该整还是得整起来,但是该读的文献也还是要继续读滴,谁让咱是个卑微的搬砖人呢?
总觉得纯生信快过时了,别人不还是一篇接一篇地中?就在1月17日(2022年的哦),frontier in genetics杂志发表了一篇纯生信文章《Identification of Potential Prognostic Biomarkers Associated With Macrophage M2 Infiltration in Gastric Cancer》,主要关于肿瘤预后标志物的筛选,别犹豫了,这份攻略赶快收藏起来,说不定下一个中SCI的就是你~

杂志封面
本文的数据来源非常简单,主要是GEO数据库中的3个胃癌芯片,包括GSE54129、GSE79973和GSE118916,详细信息如下图:

数据来源
如此简单的数据,作者是如何玩出花样的?且听小编细细道来~
Part 1. DEGs鉴定和功能富集分析
说起鉴定肿瘤预后标志物,懂得人都懂,第一步当然是找差异啦~毕竟有差异,才能有对比,有对比才能有意义嘛~
本文作者的研究思路亦如此,他们在GEO数据库中选取3个胃癌(GC)芯片数据(GSE54129、GSE79973和GSE118916),筛选差异表达基因(DEGs),绘制了火山图如图1A。
差异基因千千万,究竟应该怎么选?莫慌,小编告诉你一个万能又很实用的方法—“取交集”,没错!取交集就是在差异中找共性,共性才能说明问题嘛!试想一下,当所有的数据都说明一个共同的问题的时候,说服性和可靠性是不是更高呢?果不其然,作者取了3个GC芯片数据中共同表达的337个差异基因,绘制Venn图如图1B;悄悄说一句,本文中作者只选择了GEO数据库中的3个芯片进行了分析,如果想要增加文章的可靠性和丰富性,我们也是可以选择不同数据库(如TCGA等)中的数据取交集滴~
有了差异表达的基因,我们就可以放开手脚大胆干了,不出意外的话,接下来就该进行差异表达基因的富集分析了,知己知彼,才能百战不殆嘛!这个富集过程就是对这些差异表达的基因们进行一个简单的了解,主要弄明白这些差异基因在分子层面发挥的作用(分子功能,MF)、在细胞中的存在位置(细胞成分,CC)、参与的细胞过程(生物学过程,BP)以及参与的信号通路(KEGG)。富集分析结果的可视化有很多种形式(气泡图、条形图等),在本文如图1C。
图A B C,动动小手3张图一拼,这组图1不就出来了吗?忍不住夸自己真真是一个机智的小编呢!

图1
Part 2 使用CIBERSORT进行免疫细胞浸润预测
此部分在论文中是放在最后才介绍的,但是为了便于理解,小编把其调整到前面进行解读,因为本文是“在胃癌中鉴定与M2巨噬细胞浸润相关的潜在预后生物标志物”,贴心的小编怕有些迷糊的小可爱一头雾水:免疫细胞那么多,为什么单单就选择M2巨噬细胞呢?是无心之举?还是刻意为之?我们提前把话讲清楚哦,这可不是随便选选的哦,作者可是下了一番苦功夫的呢?作者使用CIBERSORT反卷积算法对免疫细胞浸润进行预测,才发现“M2巨噬细胞”在胃癌肿瘤和正常样本中是显著性差异表达的,于是才选择它作为研究对象,可真是个宝贝疙瘩儿~
Part 3. 通过WGCNA鉴定巨噬细胞相关模块和中枢基因
接下来整点儿高大上的~
首先介绍一位重磅级人物“WGCNA”,英文全称Weighted correlation network,中文名字是“加权基因共表达网络分析”,是用来描述不同样品之间基因关联模式的系统生物学方法,可以用来鉴定高度协同变化的基因集,并根据基因集的内连性和基因集与表型之间的关联鉴定候补生物标记基因或治疗靶点。相比于只关注差异表达的基因,WGCNA利用数千或近万个变化最大的基因或全部基因的信息识别感兴趣的基因集,并与表型进行显著性关联分析。一是充分利用了信息,二是把数千个基因与表型的关联转换为数个基因集与表型的关联,免去了多重假设检验校正的问题。
话不多说,接下来就上干货~
如文中所讲,M2巨噬细胞在胃癌患者和正常人之间存在统计学差异,于是作者就对M2巨噬细胞和337个DEGs进行加权基因共表达网络分析。以M2巨噬细胞作为外观性状,对GC样本进行聚类分析,如图2A所示;在构建共表达网络之前,还有一步比较关键的步骤—寻找最优软阈值,如图2B;关于寻找最优软阈值的内容,不动的小伙伴自行百度吧,很多博主讲的很详细,在这里小编就不赘述了,主要怕懂的小伙伴打我
基于最优软阈值构建共表达网络,将基因划分到不同模块后,可以绘制基因聚类树,如图2C,上半部分是基因的层次聚类树状图,下半部分是基因模块,也就是网络模块。上下对应,可以看到距离较近的基因(聚类到同一条分支)被划分到了同一模块。本文中这些差异表达的基因主要聚类为4个模块。
图2D就很简单啦,在三个数据集中,GC患者和对照组之间M2巨噬细胞百分比。
图2E是共表达模块与外部性状(M2锯齿细胞)之间的关系,横坐标为表型性状向量,纵坐标为每个模块的特征值向量,中间小格子中的数值代表每个性状和每个模块的特征值之间的相关性以及对应的pvalue。
图2F分析了M2巨噬细胞GS值和turquoise颜色模块MM值的相关性,关于GS值和MM值的解释,感兴趣的小伙伴可以去查查资料哦~不过记住一点就好,进行这一步主要是为了筛选模块中的核心基因(hub gene),在本文中鉴定了turquoise模块中的141个核心基因。

图2
Part 4. PPI网络构建和核心基因鉴定
在String数据库中对Part 1中获得的337个差异基因构建PPI网络,获得了25个核心基因。Part 3部分,通过WGCNA鉴定了turquoise模块中的141个核心基因。其中,有7个基因在这两部分中均存在,包括COL1A1、COL4A1、COL5A2、COL12A1、LUM、PDGFRB和THBS1。
Part 5.使用GEPIA和KM数据库对核心基因进行生存分析
是骡子是马?拿出来溜溜不就知道了?没错,于是作者首先利用GEPIA数据库分析这7个核心基因与GC患者的生存预后的关系,同时通过KM数据库进行验证,最终确定了COL1A1(logrank p = 8.9e−5),COL4A1(logrank p = 5.5e−07),COL12A1(logrank p = 0.002)和PDGFRB(logrank p = 8.2e−12)为GC的枢纽基因。

图3
Part 6. 枢纽基因与肿瘤纯度和免疫浸润的关系
到这里已经看到了胜利的曙光,毕竟从成千上万的基因中总算有理有据地筛选出了4个枢纽基因。但是,作者的脚步并没有从此停下,而是进一步进行了免疫浸润分析。结果发现,COL1A1、COL4A1、COL12A1和PDGFRB都与肿瘤纯度呈负相关,与CD4 T细胞,巨噬细胞,嗜中性粒细胞和树突状细胞的浸润之间存在显著相关性。

图4
Part 7. 枢纽基因的功能分析
俗话说,想要搞定女朋友,先要搞定她的闺蜜们~基因,同样也是同样的道理。想要真正了解某基因的功能,我们还要搞定它的闺蜜们,也就是相关基因,一个很好的方法就是构建一个基因相互作用网络。探索基因间相互作用和功能,除了string还有geneMANIA,geneMANIA还可以用于基因功能预测。给定一个查询基因,GeneMANIA会根据基因与它的相互作用,找到可能与它共享功能的基因。在本文中,鉴定出与4个枢纽基因相关的20个基因,进一步分析发现它们参与细胞外基质,细胞-基质粘附和ERBB信号通路。为了进一步探索GC中枢纽基因的功能,作者对TCGA-STAD RNA-seq数据进行了GSEA,结果发现COL1A1、COL4A1、COL12A1和PDGFRB,都富集在MAPK和PI3K-Akt信号通路中,这些信号通路与肿瘤细胞增殖、侵袭和细胞周期密切相关。
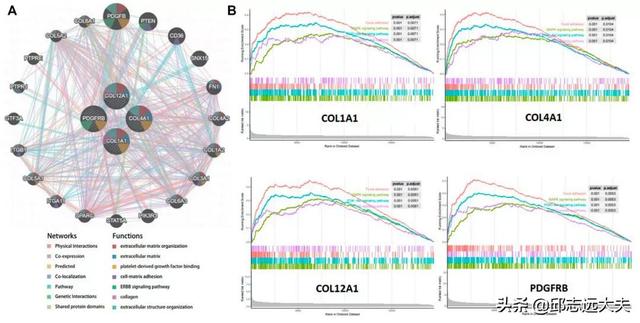
图5
结语
最后,我们再来回顾一下,整篇文章的研究思路,顺便把流程图奉上~
客观地讲,生信分析的常见套路和方法也就几种,想要玩出新意还是挺困难的,看上去相差无几的图片,总是给人一种灌水文、烂大街的感觉。纵观本文,把任何一部分单独拎出来,都可以说是毫无新意可言,并且很多图都是在线网站就可以直接生成的,也没有体现出独特的代码功力,更要命的是,数据来源也仅仅是GEO一个数据库,但是在纯生信文章接收频频遇冷的情况下,为什么作者的这篇文章还可以中呢?
小编认为,可能因为以下几点:
1)研究目标明确:全文围绕M2巨噬细胞展开,避免了做生信分析最容易犯的“东一棒槌西一榔头”的毛病,
2)采用了一些看上去比较高大上的算法:如CIBERSORT反卷积算法、加权基因共表达网络分析等;
3)多种分析手段互相验证:如GEPIA和KM数据库、string和geneMANIA数据库、常规差异表达基因和WGCNA同时鉴定核心基因等;
看到这里,一份完整的攻略已经双手奉上,还劳烦小可爱们动动手指,点个赞,可否?
参考文献:Liu, B.; Ma, X.; Ha, W., Identification of Potential Prognostic Biomarkers Associated With Macrophage M2 Infiltration in Gastric Cancer. Frontiers in Genetics 2022, 12
,




