本文由brainnews脑科学世界团队原创编译,转载请联系授权。
作为一种蛋白性疾病,阿尔茨海默病(AD)的发病机制仍有许多未解之谜。因此,科学家们尝试利用基因组、转录组、蛋白质组和代谢组水平的无偏见的数据分析来进一步了解AD的发病机制。
在2022年2月发表的这项研究中,研究者们使用串联质谱标签技术(TMT-MS),分析了1000多个AD脑组织的蛋白质组学,揭示了新的与AD相关的蛋白表达模块,这些模块在不同细胞群和脑区中高度保留。然而,其中近一半的蛋白表达模块,包括在AD中明显改变的模块,其RNA谱中却没有被观察到类似的变化。
作者特别关注了蛋白质组中的两个与AD相关的模块:一个与MAPK信号通路和代谢相关,一个与matrisome(指细胞外基质(ECM)相关蛋白的集合)相关。作者还发现,matrisome模块受APOE ε4等位基因的影响,但在调整神经病理学后发现它与认知能力下降的速度无关,而MAPK/代谢模块则与认知能力下降的速度密切相关。
解析疾病相关的蛋白组学模块或能为AD提供可靠的生物标志物,并为治疗带来希望。而且多组学联合分析,无疑能更好地理解在AD背景下这些不同数据类型之间的关系。

1.TMT识别的AD蛋白共表达网络
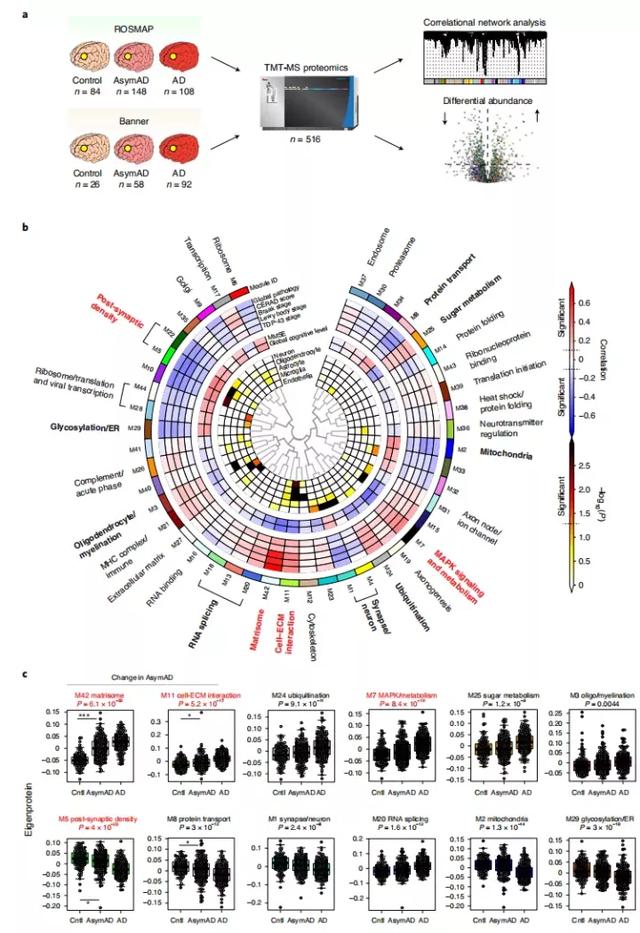
在这项研究中,作者用TMT-MS定量蛋白质组学分析了来自对照脑、无症状AD(AsymAD)脑和AD脑的总共516个背外侧前额叶皮层(DLPFC)组织。
其中,AD脑的界定是通过评定Aβ和tau神经纤维缠结沉积的半定量组织病理学以及患者在接近死亡时的认知功能。AsymAD是指有与AD病例相似的Aβ斑块和tau缠结的神经病理表现,但直至死亡时都没有明显的认知功能障碍的人群,被认为是处于AD的早期临床前阶段。
经过数据处理和剔除离群值后,使用加权基因共表达网络分析算法(WGCNA),共纳入8619个蛋白质,构建了一个包含44个模块的蛋白质共表达网络,并用GO分析来划分其生物学功能。
作者观察到这44个模块中的很大一部分与AD的至少一种病理表现或与认知功能显著相关。突触后密度、糖基化/内质网(ER)、少突胶质细胞/髓鞘化、RNA剪接、matrisome、细胞-ECM相互作用、突触/神经元、泛素化、丝裂原激活蛋白激酶(MAPK)信号传导和代谢、线粒体、糖代谢和蛋白质运输等12个模块与AD相关性比其他模块更强。
其中,模块突触后密度、MAPK信号传导和代谢、细胞-ECM互动、matrisome是与AD的神经病理症状或认知关系最密切的。与tau微管结合域(MTBR)水平升高相关性最强的模块是matrisome和cell-ECM,与MTBR负相关的模块是突触后密度和突触/神经元模块。与MTBR相关的模块在AsymAD中也倾向于改变。
为了评估一个特定的网络模块是否在AD的早期阶段发生改变,作者还比较了对照组、AsymAD和AD患者的蛋白模块。与对照组相比,12个与AD高度相关的模块中仅有4个在AsymAD中有明显增加或减少:在AsymAD中增加的模块包括matrisome和cell-ECM intERaction,而在AsymAD中减少的模块是post-synaptic density和protein transport。
总之,通过用TMT-MS技术对500多个脑组织进行分析,作者对8600多个蛋白质进行量化,并构建一个强大的蛋白质共表达网络,检测到之前未被报道的与AD相关的蛋白模块改变。有些新模块也在AD的早期阶段发生了改变,可能反映了在AD神经病理学存在的情况下但在认知能力下降之前的病理生理过程,并与tau平衡不良相关。
2.TMT AD蛋白网络包含与AD相关的模块,但在转录组中不存在
到目前为止,大多数AD的表达网络分析都是使用定量RNA测序(RNA-seq)数据进行的。然而,并非所有的mRNA转录物都与蛋白质水平有很好的相关性。
作者用来自532个DLPFC组织中测量的15582个转录数据,生成了一个AD的 RNA表达网络,通过比较这个RNA网络和从TMT-MS得到的蛋白质网络来了解RNA和蛋白质表达异同。
作者发现,RNA网络中有168个与TMT AD蛋白网络相重叠。其中,略多于一半的蛋白质模块在RNA网络中被保留了下来(44个模块中的26个),其中与AD相关的模块有突触/神经元、线粒体、寡头/髓鞘化、突触后密度、蛋白运输、细胞-ECM相互作用、RNA剪接、糖代谢。
还有18个蛋白网络模块在RNA网络中没有保留,包括与AD相关的模块 MAPK/代谢、泛素化、糖基化/ER等。在这些模块中,MAPK/代谢模块保留在RNA网络中的最少,说明其在蛋白质组中的高度独特性。
总之,作者发现蛋白质网络模块与认知功能的相关性比RNA网络模块更强,但在大多数情况下,它们与病理学的相关性与RNA模块相似。有些RNA网络模块,如MAPK/代谢模块则与相应蛋白质模块的表现相反,这或是因为在RNA水平上发生了补偿性调节。
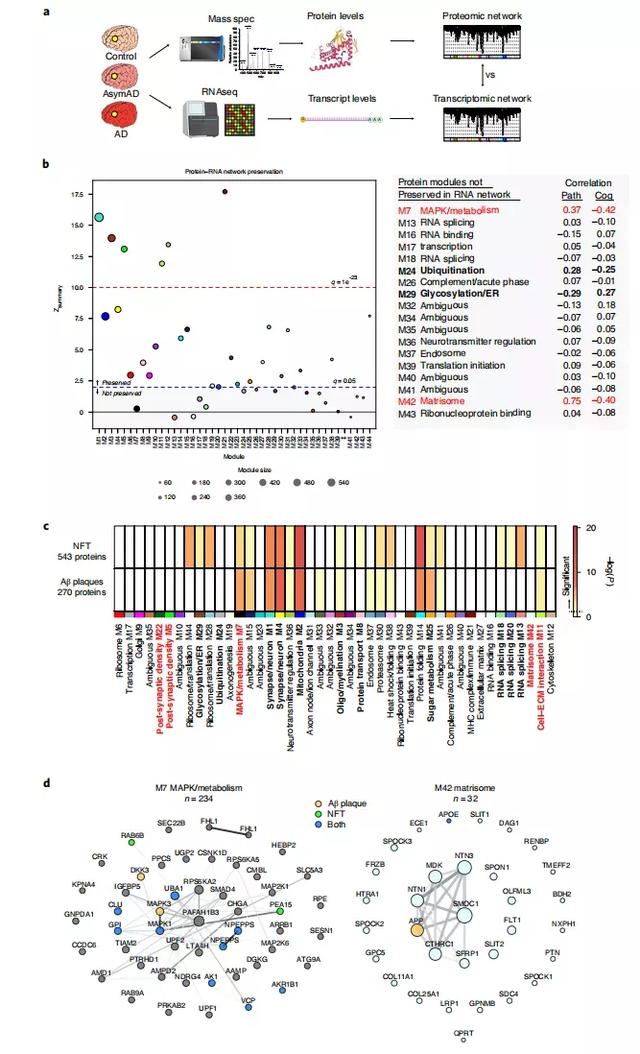
为了更好地了解从TMT-MS得到的AD蛋白网络模块与Aβ斑块和神经纤维缠结(NFTs)的空间关系,作者用激光捕获显微切割(LCM)和LFQ蛋白质组分析的方法对与Aβ斑块和NFTs共同定位的蛋白质进行了模块重叠测试,并发现模块突触/神经元、线粒体、蛋白折叠等,在斑块和缠结中都高度富集。
MAPK/代谢模块也在斑块和缠结中富集,但在Aβ斑块中的富集度更高。糖代谢模块的情况也是如此。核糖体/翻译、糖基化/ER、RNA剪接模块在NFTs中有独特的富集。然而,在LCM鉴定的核心斑块蛋白中,并没有matrisome模块的明显富集,尽管淀粉样前体蛋白(APP)和脂蛋白E(ApoE)是该模块的成员。
总之,作者发现了几个富集于NFTs和Aβ斑块中的TMT AD蛋白网络模块,如MAPK/代谢模块,与标志性的AD症状之间存在着空间关系一致性。
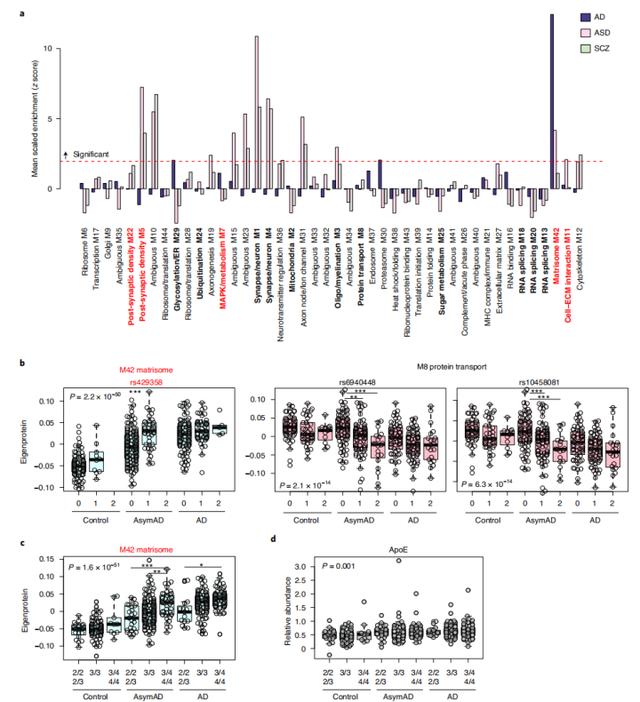
3.Matrisome模块在AD遗传风险中富集并受APOE ε4的影响
通过全基因组关联研究(GWAS),作者又评估了TMT AD蛋白模块与AD相关的遗传位点之间的关联。发现模块matrisome、蛋白酶组、糖基化/ER等与AD风险基因有明显的富集,而MAPK/代谢模块则仅表现出富集趋势。而模块matrisome的强烈富集是由该模块中的载脂蛋白驱动的,因为它对AD风险的影响很大。
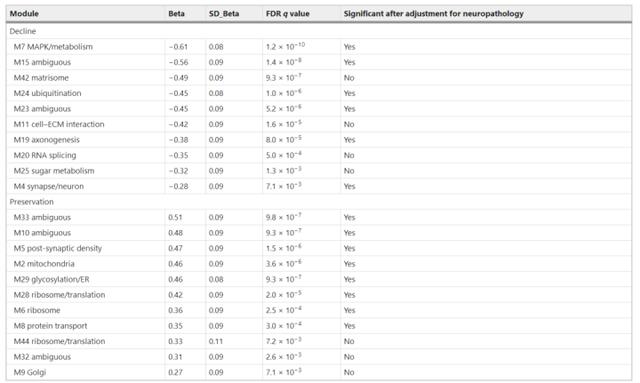
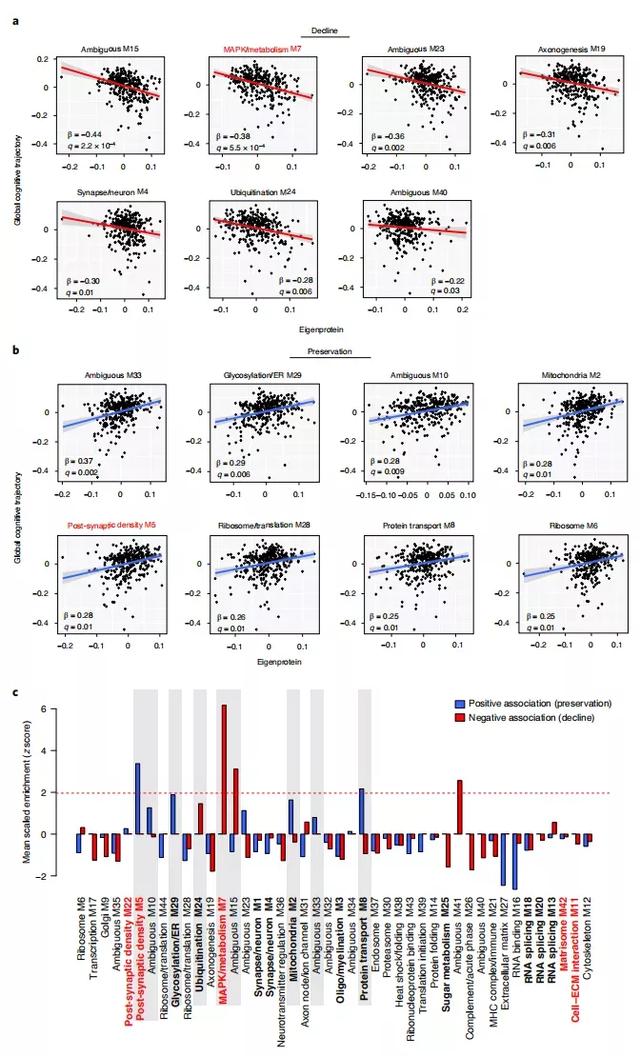
4.MAPK/代谢模块与认知轨迹有关
作者对TMT-MS AD网络中的模块蛋白水平与患者的个人认知轨迹之间的关联进行了建模,并对神经病理学进行了调整。并将调整神经病理学后仍与认知轨迹明显相关的模块列表如下。其中,与认知能力下降最密切相关的模块包括MAPK/代谢、matrisome等。【注:与认知轨迹有负相关的模块被定义为参与认知衰退的模块,而有正相关的模块被定义为参与认知保持的模块。】
总 结
综上所述,作者通过TMT-MS分析了多个中心、队列和脑区的1000多个脑组织,开发了一个强大的TMT AD蛋白网络,并利用多组学方法,发现了与AD密切相关的蛋白质网络模块,有些模块在基于RNA-seq的网络中是不存在的。这些发现强调了扩展蛋白质组分析深度、挖掘与AD相关的蛋白质共表达网络的重要性。
参考文献
Johnson, E.C.B., Carter, E.K., Dammer, E.B. et al. Large-scale deep multi-layer analysis of Alzheimer’s disease brain reveals strong proteomic disease-related changes not observed at the RNA level. Nat Neurosci 25, 213–225 (2022). https://doi.org/10.1038/s41593-021-00999-y
编译作者:米佳(brainnews创作团队)
校审:Simon(brainnews编辑部)
,




