今日推送的文章是发表在Journal of Biological Chemistry的“Structures of Streptococcus pyogenes Class A sortase in complex with substrate and product mimics provide key details of target recognition”,作者为西华盛顿大学化学系的D. Alex Johnson。

细胞壁是细菌和许多其他生物的一个关键的细胞外屏障。在细菌中,这一结构层由肽聚糖组成,它保持细胞的形状和结构的完整性,并为显示各种蛋白质因子提供支架。革兰氏阳性菌利用分选酶,这是一种半胱氨酸转肽酶,它识别并切割特定的分选信号,然后将含有分选信号的蛋白质连接到肽聚糖前体脂质II,可使得蛋白质附着在细胞壁上,。
排序酶在革兰氏阳性细菌中广泛存在,目前被分为多个类别(A- f),包括几种被认为是一般内部性的酶,以及那些聚集菌毛的酶(C类)。排序酶的机制涉及两个催化步骤:i.识别和切割目标序列,形成酰基酶中间体; ii.亲核攻击第二个反应物,初始化一个连接创建一个新的肽键的反应。关于排序酶结构和机制的大部分知识都集中在A类排序酶上。这些结构揭示了排序酶共享一个保守的核心反平行结构,称为排序酶折叠。文章试图通过对化脓性链球菌分类酶A (spySrtA)催化机制中多种状态的结构表征来填补对SrtA目标识别的认识上的空白(图1)。

图1
肽结合的spySrtA结晶和结构测定
spySrtA的大多数预测和验证的体内靶点都具有LPXTG底物序列。spySrtA在体外很容易接受LPXTA和LPXTS底物,尽管事实上这些特定的序列变异似乎不存在于体内自然发生的spySrtA底物中。spySrtA识别非甘氨酸亲核试剂和接受P1 位置不同底物的能力与saSrtA形成了鲜明的对比,saSrtA在这些位点上对甘氨酸有狭窄的选择性。
而后作者表达并纯化了含有失活C208A突变的spySrtA蛋白,并通过SDS-PAGE和LC-ESI-MS对其进行分析。我们成功地结晶和解决了C208A spySrtA结合到模型肽(P1′位置,粗体)Abz-LPATAGK(Dnp)-NH2和Ac-LPATSG-NH2的两个结构(图2A)。

图2
将溶解的酶-底物复合物称为spySrtA-LPATA和spySrtA-LPATS(图2B)。一般来说,晶体在传统的冷冻溶液中生长堆积,相对不稳定。在非对称单元中含有2个原聚体。低温条件的优化,即使用peg400作为低温保护剂,导致更好的质量的晶体衍射数据用于解决spySrtA-LPATS结构。该晶体衍射到1.4 Å分辨率,得到的结构在空间群p21中求解到Rwork/Rfree = 0.17/0.19,两个spySrtA分子在不对称单元中。spySrtA- lpats和apo spySrtA之间的单位胞和空间群非常相似。将spySrtA-LPATA链(或原体)与spySrtA-LPATA的两个分子比对,显示出非常相似的结构,两种结构的所有原体的主链原子的成对RMSD值均<0.13 Å。作者模拟spySrtA-LPATA原体B和spySrtA-LPATA中该酶的所有残基,显示出一个此前未在apo结构中看到的额外n端螺旋。
spySrtA目标识别的立体化学研究
看到清晰的肽密度,并模拟了所有spySrtA原体的整个五肽基序(图2B,C)。通过省略多肽原子并进行一轮精化得到的无偏电子密度图,证实了多肽残基的强电子密度。将spySrtA- lpats与apo spySrtA的两个(A和B)原聚体进行比对,主链原子的RMSD值分别为:0.158 Å(508个原子)和0.189 Å(541个原子)。这些结构之间最大的区别是β7-β8 3、β7-β8 4和β7-β8 5环残基的主干大约有1 Å的位移(图2D)。在这里,上标编号指的是相对于催化C208残基的残基位置。这表明,为了容纳目标肽,需要非常小的结构重排。在spySrtA-LPATA的原聚体,我们看到Abz的2-氨基和P188的羰基之间有一个潜在的氢键。作者不认为这种相互作用对该底物的结合是关键的,因为在spySrtA-LPATA复合物的b原体中没有观察到这种相互作用。Ac-LPATSG-NH2肽的成功结合和共结晶进一步支持了这一点,该肽缺乏Abz单元。
文章分析了CWSS的LPATX基序中的位置特异性相互作用。P4高度保守的Leu残基与β6-β7环路的V186、V191和V193以及β7中的V206和β8中的I218形成的疏水口袋相互作用(图3A)。P3中的脯氨酸残基通过与V206和A140、β4和β7链的残基以及β3-β4环中的M125的范德华互作弱相互作用(图3A)。这些残基之间的距离与saSrtA-LPAT*结构中的距离相等或更短,在saSrtA-LPAT*结构中观察到强烈的分子间核Overhauser效应(NOEs)。
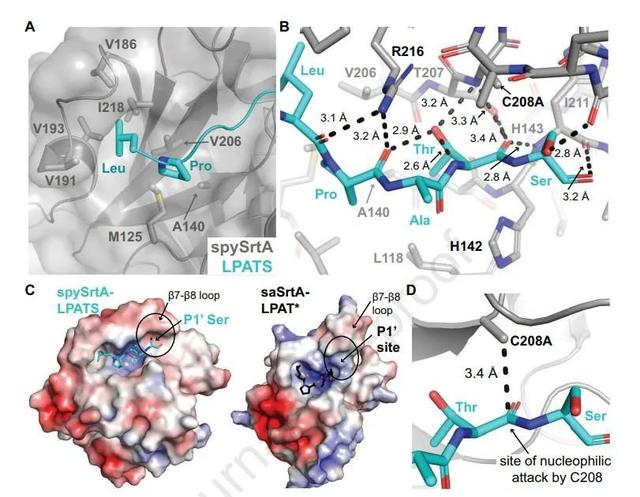
图3
在LPATX基序中有几个主链原子与spySrtA中的残基形成非共价相互作用(图3B)。在LPATA和LPATS结构中,P4 Leu和P3 Pro残基的羰基氧与R216(催化精氨酸残基)的氮原子形成氢键。在LPATA配合物中,R216也与P2 Ala羰基相互作用,而在LPATS结构中,该羰基旋转180º,并与溶剂相互作用(图3B)。与saSrtA-LPAT*结构相比,在所有结构中,观察到的P2和P1残基侧链(AT)的取向分别旋转了180º。在spySrtA-LPATA和spySrtA-LPATS中观察到的构象被称为“thrin”,以描述指向酶的P1苏氨酸侧链。P1苏氨酸的羰基氧进一步与C208A的酰胺和T207的侧链羟基以及H143的酰胺相互作用,残渣立即与催化组氨酸H142的c端相互作用(图3B)。P1 Thr的甲基指向A140和V206的侧链原子,以及侧链原子羟基与C208A催化残基的酰胺相互作用,并与自身的酰胺和P3 Pro的羰基形成肽内氢键(图3B)。最后,spySrtA-LPATS中的P1’Ser与β7-β8环形成的弱负脊相互作用 (图3C)。在先前报道的saSrtA-LPAT*结构中预测了空间上类似的P1 '结合位点,尽管在形态和总体电荷上存在一些差异(图3C)。在我们的spySrtA-LPATS结构中,我们还观察到与P1’Ser的羟基和I211的羰基形成氢键(图3B)。这种相互作用在spySrtA-LPATA复合物中必然是不存在的,因此认为它不是底物结合的必要条件。总的来说,观察到的P1 ' Ser和相邻的P1 Thr的位置使LPATS肽处于催化半胱氨酸残基亲核攻击的理想位置。其中,spySrtA-LPATS结构中C208A的甲基与P1 Thr羰基碳的距离为3.4 Å (图3D),P1-P1 '连接的剪刀式酰胺键也位于催化组氨酸(His142)附近。
酰基酶中间体模型
使用我们的spySrtA-LPATS复合物结构来模拟酰基酶中间体(图4A)。作者确定了裂解肽的坐标,并将其与spySrtA-LPATS的实验电子密度相吻合。此外,在硅胶中将C208A突变为野生型半胱氨酸,并进行一轮优化以验证肽结构,对酰基酶模型进行了最快速的能量下降最小化,以获得最终的几何形状。得到的酰基酶模型与spySrtA-LPATS结构非常相似,包括LPATS底物的P4-P2残基几乎相同的位置(图4B)。然而,在P1苏氨酸残基的情况下观察到轻微的差异。如上所述,spySrtA-LPATS中的P1 Thr羰基似乎被H143和C208A的酰胺以及T207的侧链羟基稳定。这些相互作用在作者的模型中基本维持了下来,但是观察到P1羰基向T207轻微旋转(图4B)。具体来说,在酰基-酶模型的几何结构中,P1 Thr羰基上的T207羟基为3.3 Å(图2),该距离在spySrtA-LPATS求解结构中为3.4 Å(图3B)。

图4
在催化机理方面,spySrtA-LPATA和spySrtA-LPATA结构所共有的酰基酶模型的一个特点是P1 Thr羰基与假定的催化精氨酸(R216)侧链之间没有明显的相互作用。
这是很重要的,因为这种精氨酸被认为可以稳定在分选酶连接反应中产生的高能氧阴离子中间体。在我们的酰基酶模型和解决的结构中,P1 Thr羰基实际上指向远离R216侧链,这些位点之间的距离为>6 Å(图4C)。尽管如此,研究发现R216对spySrtA的功能至关重要,因为当用模型LPATG/S/A肽底物测试时,将其突变为Ala残基会导致酶活性完全丧失(图4D)。
在氧阴离子稳定方面,作者的结构更符合T207侧链羟基的关键作用。这个残基,连同H143和C208的酰胺,处于理想位置,可以与P1 Thr羰基结合,并有可能稳定在酰基酶状态之前和亲核攻击之后形成的四面体氧阴离子中间体脂质II(图4C)。在催化Cys之前的Thr的这种类型的作用确实在以前的计算研究中被提出。此外,NCBI数据库中400个排序酶A的序列分析显示,超过90%(363个)的苏氨酸残基位于催化Cys之前,这表明该苏氨酸残基在关键反应中间体的稳定等方面起着至关重要的作用。与这一假设一致的是,我们发现spySrtA的T207A突变体几乎完全丧失了酶活性(图4D)。
spySrtA与类脂II结合的结构和生化分析
为了可视化spySrtA与脂质II的相互作用及其相关连接产物,作者合成了一个模型分枝肽,代表了LPATX底物与化脓性链球菌脂质II的肽间桥/五肽干部分的连接(图5A)。作为LPAT-LII模型是否被酶识别的初步评估,作者将其用于一个spysrta催化的模型反应中,发现它在Thr和Ala残基之间的预期位置有效切割。事实上,作者发现LPAT-LII比相关的Abz-LPATAGK(Dnp)-NH2肽反应更快,这表明添加的肽间桥/五肽干部分可能增强了spySrtA的结合和识别。总的来说,spySrtA的结构非常相似,主链原子的排列RMSD = 0.082 Å(559个原子)。在spySrtA-LPAT-LII“thrin”结构中,LPATA部分的立体化学与肽结合结构一致。spySrtA-LPATA的A-原聚体和b -原聚体的主链原子排列RMSD分别为0.218 Å(518个原子)和0.205 Å(495)。肽间桥二氨酸和l -赖氨酸残基ε-amine/ε-carbon的位置也很好地保存在“thrin”和“throut”结构之间(图5D中的灰色箭头)。这些位点与β7-β8环的残基接触,并似乎被spySrtA中由4个氨基酸形成的疏水囊所稳定(图5E)。除了l -赖氨酸的ε-碳外,在两种结构之间的五肽干的构象有更多的可变性;这反映了这些残基较弱的电子密度(图5C-D)。事实上,在这两种结构中,只有一种观察到与脂质II五肽和spySrtA酶的非共价相互作用,即在spySrtAα1 Y120羟基和脂质II d -异谷氨酰胺残基之间形成氢键。在每一个中,也存在与对称性相关分子的脂质II五肽和spySrtA酶的多重相互作用。
综上所述,研究结果表明,虽然肽间桥可能在SrtA对脂质II的识别中发挥重要作用,但五肽干并没有与酶发生实质上的相互作用。五肽柄的电子密度弱于LPAT段和肽间桥二氨酸,表明LPAT- lii配体的柄区具有灵活性(图5C)。二氨酸肽间桥的明确电子密度揭示了一个离散的结合位点,该位点可能与标准LPXTG底物基元之外的底物结合有关,特别是在P2 '位置。
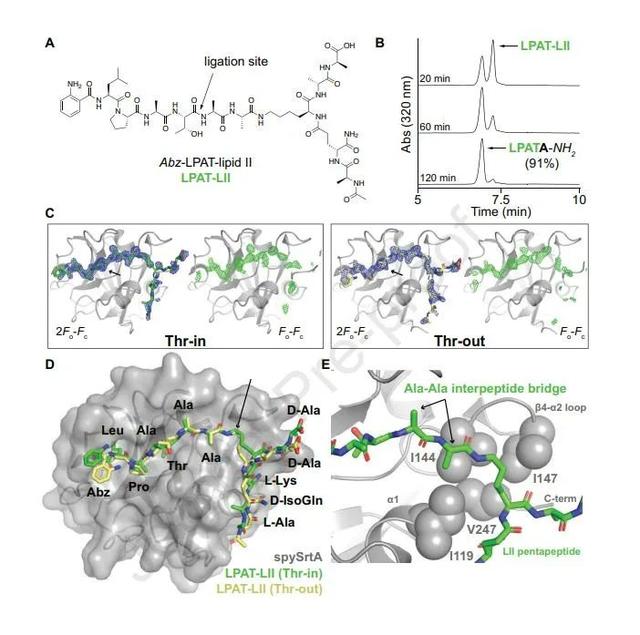
图5
spySrtA与目标肽结合的分子动力学模拟
在spySrtA-LPAT-LII的结构细化和模型构建过程中,我们观察到五肽干的电子密度比LPAT序列和肽间桥降低,这表明LPAT- lii配体不同片段的构象动力学存在变化(图5C)。对apo spySrtA (PDB ID 3FN5)、spySrtA- lpata、spySrtA- lpats和spySrtA- lii“thrin”结构进行了900纳秒的分子动力学模拟。简单地说,利用AMBER99SB*-ILDN力场对显式水进行全原子细节的MD模拟。
作者发现spySrtA的保守Arg (R216)对酶的功能至关重要(图4D,图S4E),并且通过直接接触P4和P3羰基对LPXTX基序的定位起作用(图3B),但我们没有发现与P1羰基相互作用的证据。事实上,配合物中的P1羰基远离R216侧链,相反与一系列其他位点形成相互作用(图3B,图4C),包括与活性位点Cys (C208)相邻的保守Thr残基(T207)的羟基。这些相同的接触似乎也为稳定高能量反应中间体提供了合适的氧阴离子孔,我们发现spySrtA的T207A突变体基本上不活跃,这支持了这一发现(图4D)。在这种酶的保守Arg野生型saSrtA突变为Ser,但它仍然是一个有效的转肽酶。虽然这种高度突变的saSrtA变体可能获得了一系列代偿性突变,从而否定了精氨酸稳定高能氧阴离子中间体的必要性,但作者认为,另一种解释是,野生型精氨酸对于创建氧阴离子孔不是关键的,它的主要功能是结合底物和控制底物选择性。因此,lmvgg特异性的saSrtA变体中的Arg到Ser突变被理解为促进了底物选择性的改变,而不是表示催化机制的根本改变。LPXTX基序外的底物部分可以与spySrtA酶进行特定的接触。因此,对这些位置如何影响体外酶活性的系统探索可能有助于使用spySrtA和其他A类排序酶优化SML。
总之,本研究报告了spySrtA结合LPXTX底物的第一个晶体结构,以及涉及脂质II的体内结扎产物模型。这些结构揭示了细菌分类酶识别底物的新细节,可能证明分类酶作为蛋白质工程工具的使用是有价值的。

END
,