
antiSMASH 可以对细菌、真菌、植物基因组中的次级代谢物生物合成基因簇进行全基因组范围内的快速鉴定,注释和分析。antiSMASH 既是一个数据库,也可以提供在线分析查询功能,也提供来配套的注释软件。今天在这里介绍网页版(V 5.1.2)的使用。
目前支持细菌、真菌、植物的序列的次生代谢物基因预测。

细菌版本:
https://antismash.secondarymetabolites.org/#!/start
真菌版本:
https://fungismash.secondarymetabolites.org/#!/start
植物版本:
http://plantismash.secondarymetabolites.org/
不同版本使用方法类似,下面是细菌版本的使用说明
使用教程在线分析可以通过2种方式输入分析数据:1. 上传本地序列文件,2. 直接通过输入NCBI 的 accession ID 。
1. 上传本地文件

- 支持文件格式:Sequence file (GenBank / EMBL / FASTA format)。注意:文件后缀 “fa” or “fasta” or “gbk” 等, "faa" 不会识别,需要修改后缀。
- 目前小编测试测试可行的数据有:
- 单菌 cds fasta (时间比用全基因组还要久)
- 单菌 全基因组序列
- 宏基因组 gene 集的 cds fasta
注意:蛋白序列,无论是否添加gff 都会报如下的错,以前倒是只能用蛋白序列的。

- 用 cds 和全基因组的结果有一丢丢差异,可自行判断哪个版本更加符合自己项目。
用 cds 序列测试的结果:

用全基因组序列测试的结果:
2. 用 NCBI accession ID
输入 NCBI 的编号

“提交”之后,进入任务状态页面,任务结束,上界面填写的邮箱会收到邮件。
任务完成--点击“result” 查看结果

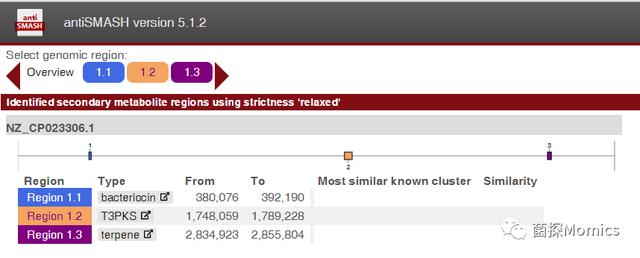
1. 结果总览界面
示例基因组,含有3个次级代谢物基因簇(Region)

界面说明:
面板A :预测到的基因簇在基因组的大概位置
面板B
Region:标签
Type :代谢物类型
From/To :基因组位置
Most similar known cluster :相似的已知代谢簇
Similarity:相似度
2. 单个基因簇结果

- G0
该基因簇所含有的基因及其分类说明
双击其中一个基因,右侧会显示具体的基因信息
- G1
上述选择的基因在基因组的位置信息
- G2
上述选择的基因比对信息
- G3
上述选择的基因结构域的信息
- G4
与该基因簇相似的基因簇,“All” 显示全部,下拉框可以只看你感兴趣的,“Download graphic” 可以下载比对结果。
,




