miRGen数据库使用教程
更多数据库干货知识
私信我回复“数据库”即可免费领取哦
亲爱的小伙伴们大家好,欢迎大家来到数据库专栏~本周我们继续讨论一下miRNA和转录因子的数据库。上周我们介绍了Transmir数据库,用来查询转录因子下游的靶miRNA或者已知miRNA查询上游的转录因子。无独有偶,还有一个Mirtrans数据库有类似的功能,总归是逃不过mir和trans哈哈,可惜的是Mirtrans数据库最近服务器有点问题不能正常使用,我们先Mark一下,下次再介绍。今天介绍的主角是DIANA Tools下的一个子数据库miRGen数据库。说到DIANA Tools下的数据库简直各个都是我的心头好啊,不仅页面简洁大气,操作起来也非常容易,各个子数据库之间长的还像,简直是学废了一个其他都可以一并学废,这种活好不粘人的数据库谁不爱呢?
miRGen数据库网址为:
http://carolina.imis.athena-innovation.gr/diana_tools/web/index.php?r=mirgenv3/index
大家使用的时候不要忘记引用参考文献哦:
Georgios Georgakilas, Ioannis S. Vlachos, Konstantinos Zagganas, Thanasis Vergoulis, Maria D. Paraskevopoulou, Ilias Kanellos, Panayiotis Tsanakas, Dimitris Dellis, Athanasios Fevgas, Theodore Dalamagas, and Artemis G. Hatzigeorgiou "DIANA-miRGen v3.0: accurate characterization of microRNA promoters and their regulators" Nucl. Acids Res. (2016) gkv1254
一、miRGen数据库功能介绍
miRGen数据库于2016年由希腊色萨利大学巴斯德研究所开发,是DIANA tool数据库网中的一个子数据库,主要用来分析miRNA的启动子和调控因子,并可批量下载。点击数据库主页面右上角的“Tools”,可链接到DIANA数据库的其他子数据库。miRGen数据库收录了7个人类细胞株(SK-N-SH, IMR90, HUVEC, HepG2, A549, K562, ESCs)和8个小鼠细胞株的结果,今年更新到了第四个版本,本次讲解以第三版为例说明。
输入网址http://carolina.imis.athena-innovation.gr/diana_tools/web/index.php?r=mirgenv3/index,进入数据库主页面:
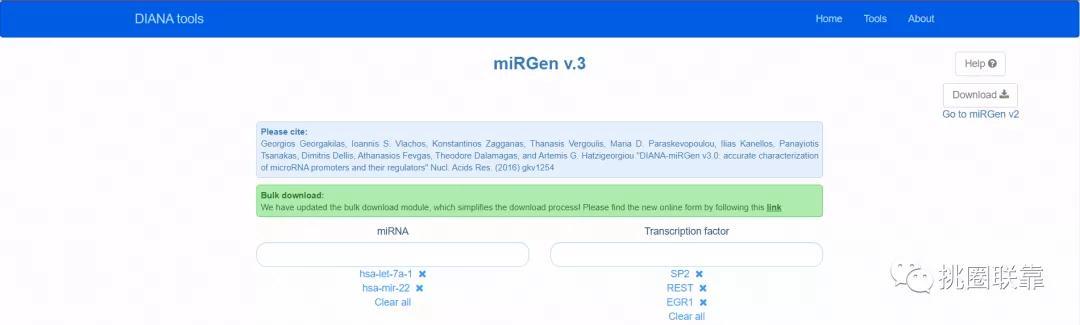
可以看到数据库中需要我们输入的一共两个地方,分别为页面中央的miRNA与Transcription factor检索框。其中在miRNA检索框中输入miRNA,即可检索可以调控该miRNA转录的转录因子,在Transcription factor中输入转录因子,即可检索该转录因子可以调控转录的miRNA。点击右上方的“Download”可以输入个人信息后请求下载miRGen数据库收录的内容。我们点击最下方的“Run our example”,以示例数据为例说明该数据库的功能:
示例数据中同时输入了miRNA(hsa-let-7a-1,hsa-mir-22)与转录因子(SP2,REST,EGR1),则结果页面展示了这两个miRNA与三个转录因子之间存在的可能的调控关系:
页面下拉,左侧为检索结果的过滤器,可以通过设置Tissues & cell lines(细胞株),Upstream & Downstream (上/下游位置),P-value(p值)以及TPM(TPM值,对,就是测序中的那个TPM,Transcripts Per Kilobase of exon model per million mapped reads)等来对结果进行筛选。右侧为对应的检索结果列表,其中miRNA name为miRBase v21收录的miRNA名称,TSS Coordinates为转录起始位点,Tissue &cell line为结果检索的细胞系/组织,DIANA Links即DIANA数据库的其他链接(mT:microT-CDS,TB: TargetBase V7, lnE: lncBASE Experimental, InP: lncBASE Predict, mP: miRPath V3)。我们以第一条hsa-let-7a-1为例,点击最右侧向下的箭头看下具体内容:
中间四行分别提供了指向miRBASE数据库、UCSC数据库、DIANA数据库的链接。下方TF name及Num of binding sites分别显示了转录因子名称和结合位点数量。我们以SP2为例,点击最右侧向下的小箭头:
出现了结合位点的具体信息,依次为motif序列,转录因子在细胞株中的表达量,Ensembl ID以及在染色体上的具体坐标等。本次示例数据是同时输入了miRNA与TF进行检索的结果,单次输入miRNA和转录因子的操作相同,此处不再赘述。
二、文章复现
我们接着结合一篇文献来看下如何使用miRGen数据库。案例文献于2018年发表在Biomedicines杂志上,影响因子为4.717分。文章的题目为Roles of NF-κB Signaling in the Regulation of miRNAs Impacting on Inflammation in Cancer,PMID:29601548:
本文为一篇综述,探讨NF-κB转录因子在肿瘤炎症中的作用,我们来尝试复现一下文章的Table 1:
结合文章内容可知,这个表主要是为了展示可以受NF-κB转录调控的miRNA有哪些。那么打开miRGen数据库后,在转录因子一栏输入NFYB。注意,在miRGen数据库中,NF-κB即为NFYB:
如上图所示,保留hsa开头的人的miRNA,并将结果整理到Excel中,即为文章中的Table 1。
好了,miRGen数据库的介绍就到这里了,大家也可以感受到这个数据库的操作属实简单,功能却十分强大。如果预测的结果比较多,大家还可以结合Transmir数据库的结果取个交集缩小一下范围。我们下期再见~
—END—
,